杏鑫娱乐 -【杏鑫精彩无限】让生活更加多彩!(籌)計算機科學與控制工程杏鑫李金豔研究團隊在國際知名學術期刊Nature Communications上發表了題為“Optimizing differential expression analysis for proteomics data via high-performing rules and ensemble inference”的研究文章。該研究為蛋白質組學數據的精准分析提供了新的視角和方法,對生物標記物和藥物靶點的發現具有重要意義🧘🏻♀️。
蛋白質組學數據的差異表達分析是生物醫學領域中的關鍵技術⟹🙅🏽,它能夠幫助科學家准確檢測特定表型的蛋白質。然而,由于分析流程中存在多種數據處理和分析工具的選擇,確定最優的分析流程一直是一個挑戰🎡。為了解決這一問題,李金豔教授領導的研究團隊對34,576種不同的差異分析流程進行了超大規模的性能評估。
通過頻繁模式挖掘新技術👱,研究團隊發現了一些能夠提高差異表達分析性能的規則,並觀察到這些規則具有跨平臺的保守屬性🦸🏿。此外𓀇,團隊還構建了分類模型🤙🏿,證明了最優分析流程的可預測性。在此基礎上👹,研究團隊設計了一種集成推斷方法🌬,整合多個高性能差異分析流程的結果,以擴大差異蛋白質組的覆蓋範圍並消除分析流程結果間的不一致性🫙。
集成推斷方法不僅顯著提高了差異表達蛋白鑒定的准確性,還促進了不同蛋白質量化方法之間的信息有效整合👨🏼🦰。例如👨👩👦👦,pAUC分數和G-均值分別獲得了高達4.61%和11.14%的提高。研究團隊還發現,使用不同蛋白質量化技術的分析流程之間具有更好的性能互補性,為設計更優異的差異表達分析方法提供了指導👚💨。
為了便于蛋白組學數據分析領域的研究者使用,研究團隊構建了網絡服務器OpDEA(http://www.ai4pro.tech:3838/)以及離線工具軟件包,以協助研究者進行差異分析流程的選擇和差異表達分析。
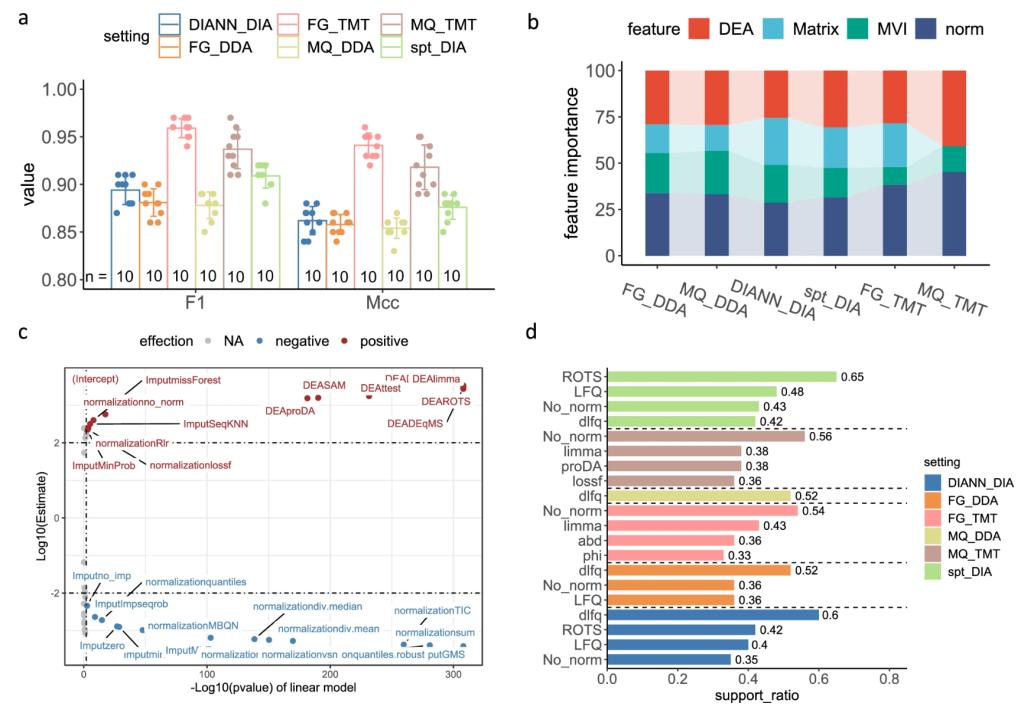
機器學習及數據挖掘技術分析差異表達分析流程的性能可預測性及保守性
閱讀原文
